在進行分類問題時,可能會碰到資料不平衡的問題。人們往往會透過模型想要找到數據中較為少數的那部分,如:信用卡盜刷紀錄、垃圾郵件識別等。當數據出現不平衡時,若模型在測試資料集中皆預測為人數較多的那個類別時,雖然可以達到較高的準確率,但並不代表此模型能夠準確幫助分類,因此在資料內數量比例超過1:4時,就建議在分析前將資料不平衡的問題納入考量。
在處理不平衡資料時,可考量使用以下幾種作法:
使用準確率以外的衡量變量:
如:精確度/召回率、F1 score等,而不再依靠準確率來作為主要的模型衡量指標
Oversampling:
將樣本數較少的組別之數據隨機選取並進行複製,使得各組之間的數據量可以達到相同,但若將同樣的數據複製多份,可能會導致模型會出現過度擬合(Overfitting)的問題。
SMOTE(Synthetic Minority Over-sampling Technique):
合成少數過採樣方法,為random oversampling改進後的方法,概念是對少數類的樣本進行分析,並人工合成新樣本添加到數據集中。由於此方法並不會考量其他樣本的情況,因此若所選取的少樣類樣本周遭皆是多數類別的樣本,則所選取到的樣本可能就是噪音,使得新合成的樣本會與多數樣本產生重疊,進而導致模型分類的困難。
Undersampling:
在樣本數較多的組別中,隨機挑選若干筆數據,使各組的數據量達到相同。此方法會丟失許多訊息,因此延伸出多個方法來減少過多的訊息量損失,如:使用Ensemble learning的方法(EasyEnsemble, BalanceCascade, NearMiss)來改善隨機抽樣導致重要樣本被刪除的問題。
調整使用的演算法
在使用的模型/演算法中將各個類別的資料數量納入考量,透過權重進行調整,此概念稱為成本敏感學習技術(Cost-Sensitive Learning)。舉例:在訓練決策樹(decision tree)時,可以設定不同權重給予不同類別,使得在訓練時,可以改善資料不平衡所遇到的問題。
在進行資料分析時,不平衡的問題常會出現,如:醫學資料中患有疾病與健康受試者常常就會出現不平衡的問題,因此此議題仍是非常重要的。
由於本次使用的資料集包含的是時序資料,而時序資料的imbalance問題有許多更加深入的處理方法,因此在本次實作中將採用模擬資料來做示範。
R:
set.seed(2022) ### 設定隨機種子,使每次產生的資料相同
n1 <- 200 # 第一類的數量(type1)
n2 <- 20 # 第二類的數量(type2)
## 產生type1的data
x <- rnorm(mean = 0, sd = 0.5, n = n1)
y <- rnorm(mean = 0, sd = 1, n = n1)
data1 <- data.frame(label=rep("type1", n1),x=x, y=y, stringsAsFactors = T)
## 產生type2的data
x2 <- rnorm(mean = 2, sd = 0.5, n = n2)
y2 <- rnorm(mean = 2, sd = 1, n = n2)
data2 <- data.frame(label=rep("type2", n2), x=x2, y=y2, stringsAsFactors = T)
## 建立 imbalanced_data
imbalanced_data <- rbind(data1, data2)
table(imbalanced_data$label)
#type1 type2
# 200 20
Python:
import random
import numpy as np
import pandas as pd
### 設定隨機種子,使每次產生的資料相同
random.seed(2022)
n1 = 200 # 第一類的數量(type1)
n2 = 20 # 第二類的數量(type2)
## 產生type1的data
x = np.random.normal(0,0.5,n1)
y = np.random.normal(0,1,n1)
data1 = pd.DataFrame({'label':['type1']*n1,'x':x,'y':y})
## 產生type2的data
x2 = np.random.normal(2,0.5,n2)
y2 = np.random.normal(2,1,n2)
data2 = pd.DataFrame({'label':['type2']*n2,'x':x2,'y':y2})
## 建立 imbalanced_data
imbalance_data = pd.concat([data1, data2])
imbalance_data['label'].value_counts()
#type1 200
#type2 20
R:
## 在較少數的類別(type2)中,隨機抽樣取後放回,產生新的資料點
new_data <- data2[sample(nrow(data2), size = n1-n2, replace = T),]
balanced_data <- rbind(imbalanced_data, new_data)
table(balanced_data$label)
#type1 type2
# 200 200
## 繪圖
library(ggplot2)
library(ggpubr)
p1 <- ggplot(data = imbalanced_data,aes(x=x,y=y,colour=label))+
geom_point()+
ggtitle("Imbalance data")
p2 <- ggplot(data = balanced_data,aes(x=x,y=y,colour=label))+
geom_point()+
ggtitle("Balanced data(Oversampling)")
ggarrange(p1,p2, ncol = 2,nrow = 1,common.legend = T,legend="bottom")
Random Oversampling所產生的點會和原始資料點重疊!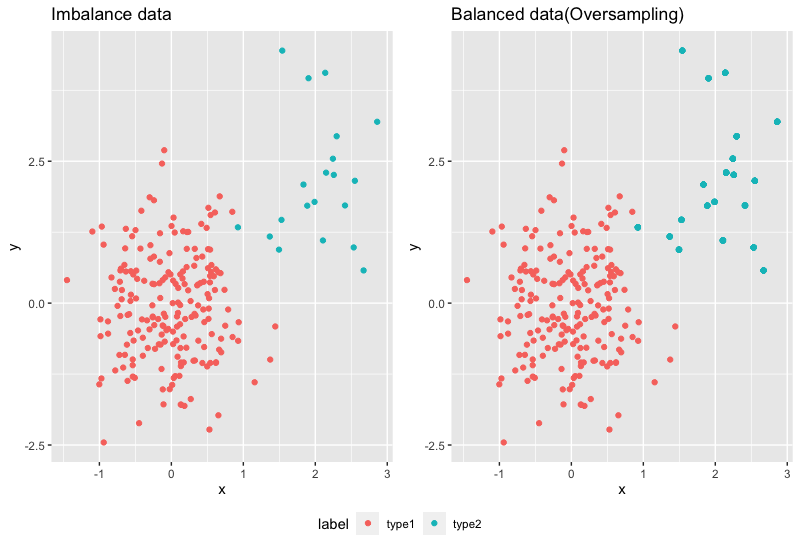
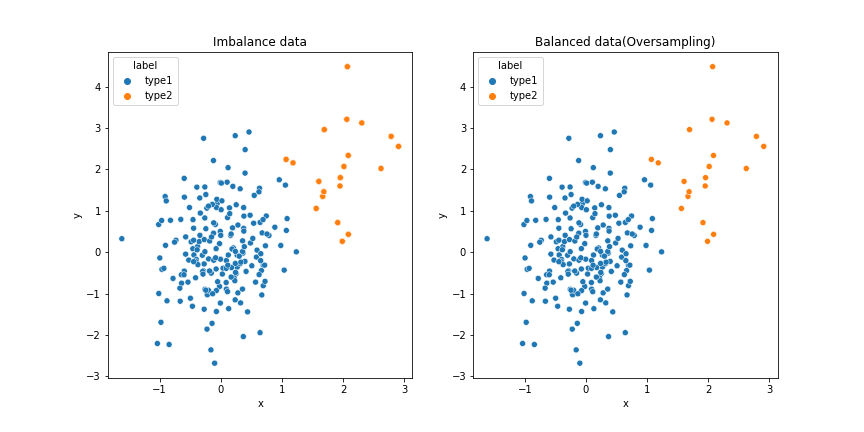
Python: 使用imblearn.over_sampling套件中的RandomOverSampler
import imblearn
from imblearn.over_sampling import RandomOverSampler
from collections import Counter
oversample = RandomOverSampler(sampling_strategy='minority')
X_over, y_over = oversample.fit_resample(imbalance_data[['x','y']], imbalance_data['label'])
balanced_data = X_over
balanced_data['label'] = label
print(Counter(balanced_data['label'] ))
# Counter({'type1': 200, 'type2': 200})
##繪圖
import seaborn as sns
import matplotlib.pyplot as plt
sns.scatterplot(data=imbalance_data, x='x', y='y', hue='label',ax=axs[0]).set(title='Imbalance data')
sns.scatterplot(data=balance_data, x='x', y='y', hue='label',ax=axs[1]).set(title='Balanced data(Oversampling)')
R: 使用performanceEstimation套件的smote語法
library(performanceEstimation)
## default: smote(form, data, perc.over = 2, k = 5, perc.under = 2)
## perc.over: 控制minority class進行over-sampling
## perc.under: 控制majority classes進行under-sampling
newData <- smote(label ~ .,imbalanced_data, perc.over = 4)
table(newData$label)
#type1 type2
# 160 100
## 繪圖
p3 <- ggplot(data = newData,aes(x=x,y=y,colour=label))+
geom_point()+
ggtitle("Balanced data(SMOTE)")
ggarrange(p1,p3, ncol = 2,nrow = 1,common.legend = T,legend="bottom")

Python: 使用imblearn.over_sampling套件中的SMOTE
from imblearn.over_sampling import SMOTE
X_smote, label = SMOTE(random_state=2022).fit_resample(imbalance_data[['x','y']], imbalance_data['label'])
newData = X_smote
newData['label'] = label
print(Counter(newData['label'] ))
#Counter({'type1': 200, 'type2': 200})
##繪圖
fig, axs = plt.subplots(ncols=2,figsize=(12, 6))
sns.scatterplot(data=imbalance_data, x='x', y='y', hue='label',ax=axs[0]).set(title='Imbalance data')
sns.scatterplot(data=newData, x='x', y='y', hue='label',ax=axs[1]).set(title='Balanced data(SMOTE)')

R:使用ROSE套件,ovun.sample語法
library(ROSE)]
# 設定method為'under', N的數量為minority class的兩倍
balanced_sample <-ovun.sample(label ~ ., data = imbalanced_data, method = "under", N=n2*2, seed = 5)$data
table(balanced_sample$label)
#type1 type2
# 20 20
## 繪圖
p3 <- ggplot(data = newData,aes(x=x,y=y,colour=label))+
geom_point()+
ggtitle("Balanced data(SMOTE)")
ggarrange(p1,p3, ncol = 2,nrow = 1,common.legend = T,legend="bottom")
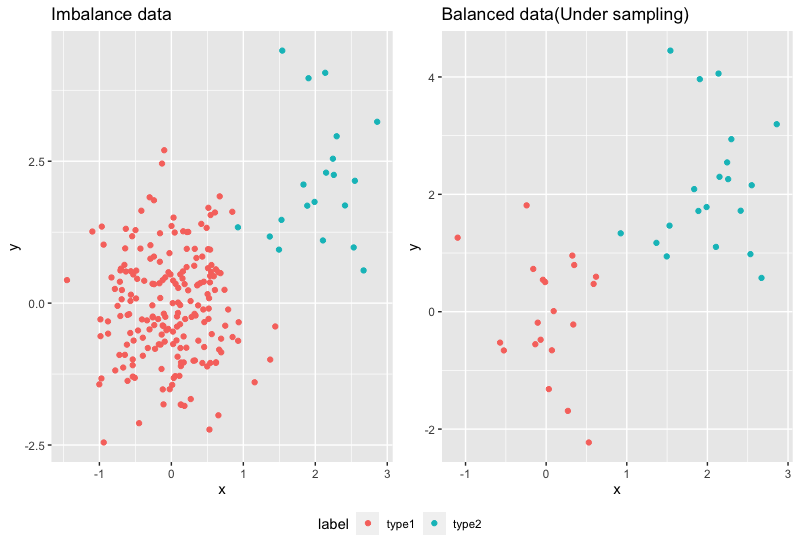
Python: 使用imblearn.under_sampling套件中的RandomUnderSampler
from imblearn.under_sampling import RandomUnderSampler
oversample = RandomUnderSampler(sampling_strategy='majority')
X_under, label = oversample.fit_resample(imbalance_data[['x','y']], imbalance_data['label'])
balanced_sample = X_under
balanced_sample['label'] = label
print(Counter(balanced_sample['label'] ))
# Counter({'type1': 20, 'type2': 20})
##繪圖
fig, axs = plt.subplots(ncols=2,figsize=(12, 6))
sns.scatterplot(data=imbalance_data, x='x', y='y', hue='label',ax=axs[0]).set(title='Imbalance data')
sns.scatterplot(data=balanced_sample, x='x', y='y', hue='label',ax=axs[1]).set(title='Balanced data(Under sampling)')

